В журнале The Journal of Chemical Physics (IF 3,1) по результатам работы, выполненной совместно с коллегами из Международного томографического центра СО РАН и Института химической биологии и фундаментальной медицины СО РАН, опубликована статья, соавтором которой является д.ф-.м.н., профессор Е.Г. Багрянская (руководитель отдела физической химии, директор НИОХ СО РАН).
Exploring combined spin-labeling approach for structural studies of mRNA in the human ribosome
Mikhail Kolokolov, Alexey Malygin, Dmitri Graifer, Mariya Meschaninova, Mariya Vorobyeva, Matvey Fedin, Olesya Krumkacheva, Elena Bagryanskaya
J. Chem. Phys.,
Volume 162, Issue 11 14 March 2025
Special Collection: Dynamic Exciton for Materials, Biology and Energy Conversion
DOI: 10.1063/5.0245722
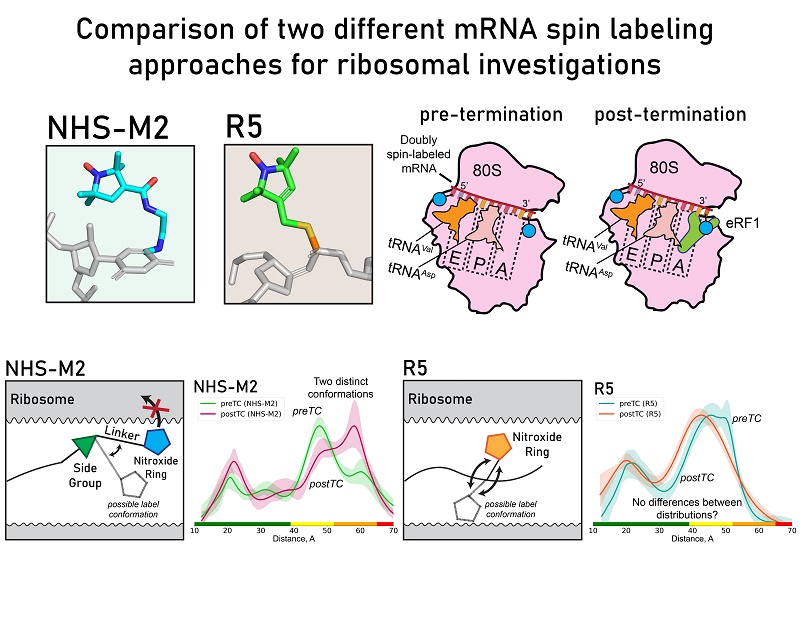
Аналог мРНК, спин - меченный по рибозофосфатному остатку, был применен для изучения структуры мРНК в рибосоме человека. Было обнаружено влияние структуры и расположения спиновых меток на измеряемые межспиновые расстояния в комплексах рибосом с мРНК. Экспериментальные результаты, полученные с использованием дипольной ЭПР-спектроскопии и расчетами молекулярной динамики, показали, что метки, введенные в нуклеотидные основания, обеспечивают более высокое разрешение между конформациями мРНК в канале мРНК рибосомы, по сравнению с метками, введенными в рибозофосфатный остаток. Разработанный новый метод введения спиновых меток может найти применение в исследованиях структуры РНК и их комплексов. Преимущества этого метода заключаются в возможности введения меток в любое положение цепи РНК, а также в использовании короткого линкера, что повышает точность определения расстояний между местами введения.
Author Notes
In this study, we investigated the structural variability of mRNA in the human ribosome by comparing two spin-labeling strategies: one involving an mRNA analog bearing two spin labels attached to the ribose-phosphate backbone and the other placing labels at the nucleotide bases. The use of two strategies of spin labeling of mRNAs allowed us to study for the first time the effect of the structure and location of spin labels on the measured interspin distances in human ribosome complexes. Experiments using dipolar EPR spectroscopy, supported by molecular dynamics calculations, demonstrated that labels introduced at nucleotide bases provide a higher resolution between mRNA conformations in the ribosome mRNA channel, compared to labels introduced at the ribose-phosphate backbone. Although ribose-phosphate labeling turned out to be less informative on its own for studying mRNA conformations in the ribosome than the previously used base labeling, it can find application in other complex studies of the structure of RNAs and their complexes.

